J’ai eu le plaisir d’avoir une entrevue avec le Dr. Julien Cohen-Adad, afin de discuter de son travail, et plus particulièrement de son article « White matter atlas of the human spinal cord with estimation of partial volume effect »1, publié en 2015 dans NeuroImage.
Le professeur Julien Cohen-Adad fait partie du laboratoire NeuroPoly à Polytechnique Montréal, et est également co-directeur de la plateforme d’imagerie de l’institut universitaire de gériatrie de Montréal. Entre ces périodes d’enseignement, il aide les chercheurs en les guidant dans la mise en place de protocoles ou en développant des outils informatiques. Les trois principaux projets auxquels il contribue actuellement sont qMRLab, une plateforme de traitement d’image, AxonDeepSeg, une plateforme permettant de segmenter des images de microscopie, basée sur l’apprentissage profond (deep learning) et Spinal Cord Toolbox. C’est dans cette toolbox, qui, comme son nom l’indique, contient des outils pour le traitement d’images de la moelle épinière, qu’est inclus l’atlas dont nous allons discuter.
Cet atlas (figure 1) permet, après avoir recalé les données d’un scan IRM, de visualiser les différentes voies de la matière blanche contenue dans la moelle épinière, tout en donnant une information sur le volume partiel des pixels, c’est-à-dire quel est la proportion de chaque voie qu’il contient.
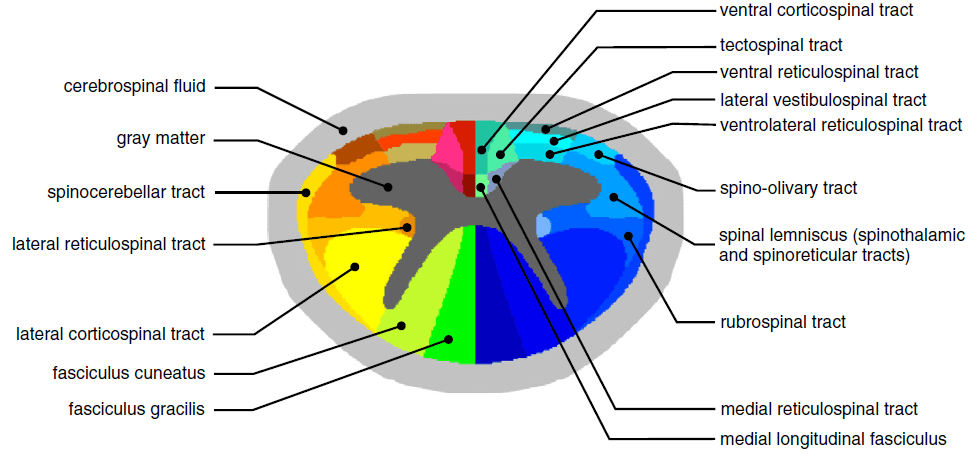
Pouvez-vous nous dire le type d’utilisation de cet atlas ?
Les utilisateurs sont composés à 50% de chercheurs, à 50% de cliniciens chercheurs et dans ces gens-là, il y a peut-être 40% d’applications sclérose en plaques, 10% d’applications sclérose latérale amyotrophique et 30% d’applications en traumatismes des compressions de la moelle épinière, par exemple des hernies ou des dégénérescences de disques. Pour le reste ce sont des sujets sains. Cet atlas est utilisé par exemple si quelqu’un étudie l’impact d’une compression de la moelle sur les voies sensorielles. Il aimerait savoir où sont les voies sensorielles dans la moelle. Pour ça, on utilise un atlas. Ce que les gens faisaient jusqu’à présent, c’est qu’ils regardaient un textbook ou Gray’s Anatomy par exemple pour y trouver un dessin qui a été fait il y a 50 ans d’une moelle épinière, et essayaient de faire la correspondance entre les pixels et puis l’atlas. L’idée de cet atlas était de digitaliser un atlas déjà existant et de le recaler de manière non-rigide dans un template qui sert également de template de recalage pour des images IRM. Typiquement, une personne va recaler un sujet sur ce template. Ce template contient l’atlas, donc la correspondance de pixels va se faire automatiquement et de manière non biaisée.
Mais n’y a-t-il pas un biais qui est créé par l’utilisation d’un atlas déjà existant ? Et est-ce que cela s’adapte bien à tous les sujets ?
Alors oui, il y a un biais de facto puisqu’on utilise un atlas déjà existant. Maintenant c’est comment limiter ce biais. En utilisant d’autres atlas. Si on utilise trois autres atlas, on aura un biais lié à ces trois atlas. Le problème est qu’il existe très peu d’atlas comme cela. Ils se comptent sur les doigts d’une main. Et les autres qui existent sont de très mauvaise qualité ou n’ont pas suffisamment d’informations sur les voies. C’est pour cela qu’en ce moment, il y a un manque de connaissances générales sur la manière dont sont distribuées les voies dans la moelle épinière. Maintenant, y a-t-il un biais par rapport à la population ? Alors oui, c’est un atlas qui a été pris sur un adulte. Si on utilise maintenant une population pédiatrique, est-ce que les voies sont distribuées de la même façon ? A ma connaissance personne dans le monde n’a cette réponse. Donc il y a de la place pour étudier ces questions-là.
Dans cet article, on propage la coupe de référence, la C4, de proche en proche pour avoir l’ensemble de la moelle épinière. Pourquoi la tranche C4 est-elle prise comme référence et comment se fait cette propagation ?
La réponse principale est que le Gray’s Anatomy, l’atlas de référence, est pris au niveau C4-C5. Une des raisons corollaires probablement est que c’est là où se trouve l’élargissement cervical. C’est là où il y a toutes les efférences motrices de nos membres supérieurs. Donc la moelle y est un peu plus grosse, un peu plus facile à étudiée aussi. Ensuite pour propager l’atlas, on utilise un algorithme de recalage qui est basé sur un article publié. On va calculer un champ de déformation régularisé, qui suit certaines contraintes, parce qu’on s’entend qu’en terme de recalage on peut recaler n’importe quoi, comme un éléphant sur un poussin. On va ensuite appliquer ce champ de déformation aux 30 masques de matière blanche, chacun correspondant à une voie de la moelle épinière.
Cela suppose qu’il y ait toujours la même proportion de chaque voie dans la moelle épinière ?
Oui, absolument. Est-ce que c’est justifié ? Encore une fois, personne dans le monde à ma connaissance n’a cette réponse.
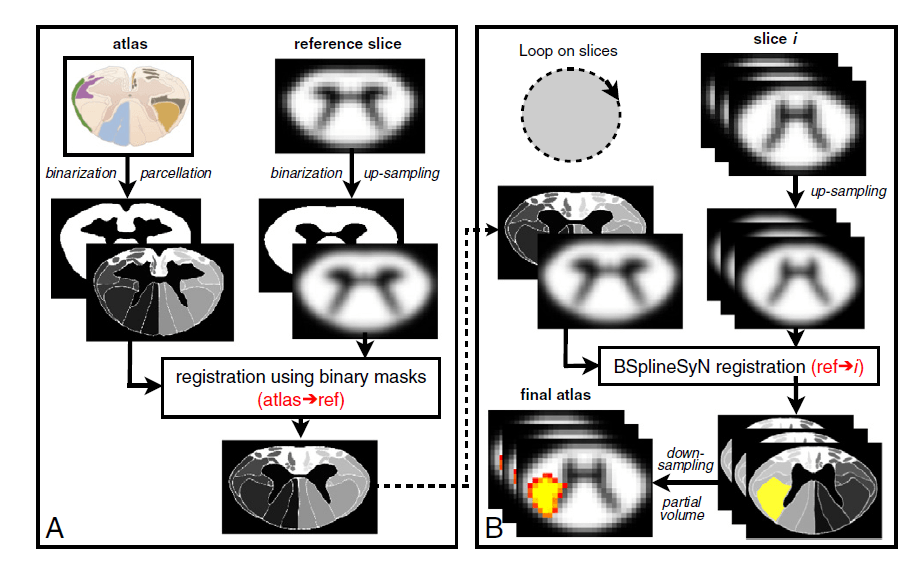
Lors du processus de création de l’atlas (figure 2), on sur-échantillonne l’atlas pour le sous-échantillonné par la suite. Quel est l’intérêt ?
En fait on avait digitalisé cet atlas du Grey’s Anatomy à très haute résolution et ensuite on l’a sous-échantillonné pour le mettre à une résolution de 0.5mm isotrope, pour deux raisons principales. La première est qu’en IRM en général, on n’a pas une résolution de 60 micromètres. La résolution est de l’ordre du millimètre ou 0.5mm. On a créé un template à une résolution de 0.5mm pour accommoder la plupart des scans IRM. On aurait pu faire du 0.1mm. On ne l’a pas fait pour des raisons de place. Pour l’instant le template prend quelque chose comme 100 Mo. Si on divise par deux la taille du pixel dans chaque dimension, ça nous fait passer à 800 Mo ce qui est plus lourd.
Avec ce sous-échantillonnage on passe à des estimations de volumes partiels c’est à dire que chaque voxel peut contenir différentes voix. Est-ce que ça a un intérêt en pratique d’avoir ce volume partiel ?
Alors pour revenir à l’exemple qu’on avait tantôt, si un patient a une compression de la moelle par exemple, ou une lésion de sclérose en plaques à un certain endroit de la moelle épinière, on aimerait savoir jusqu’à quel point ces voies sensorielles ou motrices sont affectées. Donc on aimerait une information quantitative, comme par exemple savoir que la lésion cause des dommages sur 50% des voies motrices. Pour avoir cette information de 50%, étant donné que les pixels d’IRM sont relativement gros, 1mm, 2mm dans certains cas, et peuvent donc contenir plusieurs voix, on a besoin de ce volume partiel. De manière générale, si on veut répondre à la question du pourcentage des voix qui sont affectées par la lésion, alors on a besoin de l’information de volume partielle.
Cet article date de 2015, est-ce qu’il y a eu des avancées sur l’atlas depuis ?
Alors je dirais que l’atlas en tant que tel, pas vraiment. C’est toujours le même qu’on utilise. En revanche, il y a un nouvel article qui a été fait par Duval dans NeuroImage 2019, « Axons morphometry in the human spinal cord »2. Et là ce qui a été fait, c’est qu’on a scanné une vraie moelle épinière humaine ex vivo à haute résolution en utilisant des systèmes de microscopie électronique. Grâce à cela on a pu faire un mapping des différents axones dans la moelle épinière. Cela nous a permis d’essayer de voir si cet atlas du Gray’s Anatomy est effectivement proche de la réalité ou pas. Et on a recalé dans le template et l’atlas du Spinal Cord Toolbox toute cette information sur la microstructure de la moelle épinière humaine. Donc maintenant dans Spinal Cord Toolbox, il y a, en plus du template et de l’atlas de 2015, un atlas qui vient vraiment de la microstructure à partir d’images de microscopie électronique.
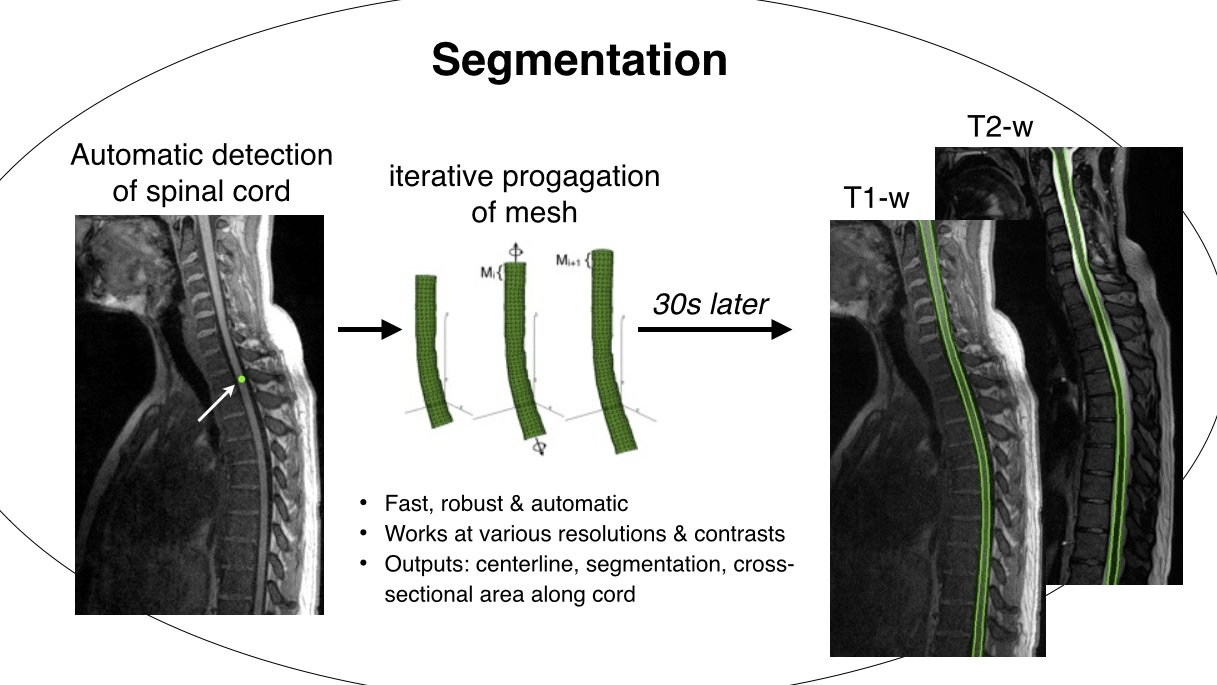
Cet atlas est disponible dans la Spinal Cord Toolbox. Qu’est-ce qu’on peut trouver d’autre dans cette toolbox ?
Plein de choses, comme par exemple un template qui est lié à cet atlas justement, des outils de recalage, par exemple de séries temporelles (figure 3). Si quelqu’un reste dans un scanner pendant 5 minutes, il y a plein d’images qui sont prises. Ces images peuvent être recalées ensemble dans le temps. Il y a également des outils de générations de rapports sous forme de pages HTML, sur la façon dont les processus se sont passés, puis d’autres features, par exemple des algorithmes de segmentation automatique de lésions de la moelle épinière ou alors des algorithmes automatiques de détection des disques intervertébraux, des choses comme ça.
Toutes ces fonctionnalités sont utilisées dans le même cadre que l’atlas ?
Alors je dirais de manière générale oui. Les gens ont une sorte de séquence de commandes qu’ils vont exécuter et qui est assez générique. Notre job ici c’est d’aider les chercheurs qui utilisent ça. Et puis parfois il y a des cas un peu outlier, où des chercheurs vont scanner une moelle épinière humaine dans un scanner très haut champ et là ils sont juste intéressés à par exemple faire un straightening, donc appliquer un algorithme qui va, si une moelle est courbée dans le scanner, l’aligner. Ils ont juste à utiliser une seule fonctionnalité de la Spinal Cord Toolbox.
______________________________________________________________________________________________________
Propos recueillis le 25 février 2019
Références
- S. Lévy, M. Benhamou, C. Naaman, P. Rainville, V. Callot, J. Cohen-Adad, “White matter atlas of the human spinal cord with estimation of partial volume effect”, NeuroImage, Volume 119, 1 October 2015, Pages 262-271
- Tanguy Duval, Ariane Saliani, Harris Nami, Antonio Nanci, Nikola Stikov, Hugues Leblond, Julien Cohen-Adad, “Axons morphometry in the human spinal cord”, NeuroImage, Volume 185, 15 January 2019, Pages 119-128