Le Dr Kadoury est professeur adjoint à l’école Polytechnique Montréal en génie biomédical à polytechnique Montréal ainsi qu’un chercheur au CRCHUM.

Il compte à son actif plus de 50 publications. Il est également titulaire de la chair de recherche du Canada en Imagerie Médicale. Il est co-auteur de l’article ‘Learning Normalized Inputs for Itérative Estimation in Medical Image Segmentation’[1] sur lequel je l’ai interrogé pour cette interview.
Il est expert en traitement d’image médical par ordinateur, en apprentissage machine et en vision par ordinateur notamment. Il cherche à développer des outils logiciels pour l’aide aux diagnostics et aux interventions chirurgicales. Il est, dans le cadre de ces recherches, rattaché au CRCHUM.
Cette article date de 2018 et est basé sur une étude lancée en 2015 grâce à un financement de MEDTEQ. Il résulte d’une étroite collaboration entre le centre de recherche du centre hospitalier de l’université de Montréal (CRCHUM) et de Polytechnique Montréal avec l’aide de l’institut de Machine Learning de Montréal (MILA) et Imagia . Ce projet est parti d’une volonté d’automatiser le processus de détection du cancer du foie grâce avec utilisation du deep learning. Ce processus s’est lentement tourné vers la segmentation des tumeurs sur les différentes images médicales. Cette article présente bien évidemment le modèle développé par le Dr kadoury et son équipe pour accomplir cette tache mais également d’autre applications comme la segmentation de la prostate sur des IRM ou encore de la segmentation en microscopie électronique. En effet, bien que la méthode fut reconnue pour sa capacitée à détecter et à segmenter les tumeurs hépatiques, elle n’en reste pas moins utile pour d’autres application. Cette article présente en somme un nouveau moyen de normaliser et de prétraiter les images biomédicale tout en montrant l’efficacité d’un tel système. L’étude se fait également un utilisant un réseaux de neurone de dernière génération qui est un fully connected Resnet afin d’effectuer la segmentation.
Le deep learning est de plus en plus utilisé pour le traitement d’image dans différents cardes d’applications notamment en classification et en segmentation (détection de contour d’objet). Cette révolution se fait également en imagerie biomédicale mais est toutefois plus complexe. En effet le Dr Kadoury nous rapporte effectivement que la quantité d’information disponible n’est à l’heure que très faible quand on la compare aux images naturelles que l’on peut trouver. La taille du Dataset à pourtant un réel impact sur la convergence et l’optimisation des réseaux de neurone et des millions de paramètres entrainables de ce dernier. On parle en effet de volumes très faibles extraits de certaines images seulement ce qui explique la faible quantité d’information que nous avons pour le moment. Ces volumes font environ deux centimètres de diamètre et donc ne se trouve pas sur toutes les images du foies qui sont récupérées lors des examens préliminaires (CTscan, IRM …).
Une centaine d’image ne représente rien face aux millions d’images disponibles pour le concours ImageNet par exemple. Ce concours aide pourtant à révéler les réseaux de neurones de dernière génération qui peuvent servir de base pour différentes applications notamment dans l’imagerie biomédicale. L’un des premier problème pour ce genre d’application est donc d’essayer de modifier le dataset ou le modèle pour pouvoir effectuer un processus d’entrainement similaire à celui effectué sur des images naturelles.
La quantité de données n’est pas la seule limitation, en effet certaines méthodes de Data augmentation peuvent permettre de passer outre ce problème, notamment en appliquant des transformations élémentaire sur les différents patchs prélevés sur les images.
Une autre limite, qui était dans le cas du Dr Kadoury beaucoup plus importante , c’est la normalisation des données. En effet, bien que les méthode pour le prise d’image soient encadré à minima, nous allons toujours récupérer une grande variabilité dans l’imagerie Médicale. Dans le cas des tumeurs hépatique cela est d’autant plus complexe que l’utilisation d’un agent de contraste est nécessaire pour rehausser la tumeur sur l’image comme le montre l’image ci-dessous [2].
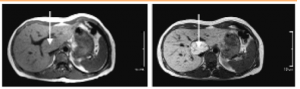
’
Le rehaussement de la tumeur est assez variable d’un patient à un autre ils ont des réponses physiologiques qui sont différentes et donc même si le timing est similaire pour la prise d’image, la réponse au niveau du rehaussement est différente’ m’a précisé le Dr Kadoury. Le résultats pourra également varier en fonction du produit de contraste comme cela est représenté dans la figure 2. De fait une certaine forme de standardisation des données est nécessaire.

J’étais assez surpris de voir ce changement dans le pipeline traditionnel et ai donc demander au Dr kadoury d’où venait cette idée. Cette découverte s’est faite en investiguant la création d’un nouveau modèle pour la segmentation basé sur FC-Resnet (gagnant du concours Imagenet en 2015). En investiguant différentes architectures ils se sont rendu compte que ‘les images sont d’une certaine manière normalisées avant de passer à la segmentation’ et ont décidé de mettre cela à profit. Une première découverte qu’ils ont effectué fut sur l’importance des skip connexion en apprentissage profond en biomédical. Cette première étude et ses résultats furent rapportés un an auparavant dans un article intitulé ‘the importance of skip connexion in biomédicale image processing’.
Dans toutes ces recherches, le focus ne s’est jamais fait sur l’état de l’image post pre-processing, mais uniquement sur l’amélioration des résultats de la segmentation. Pour mesurer ces résultats, ils ont utilisé un dataset qu’ils ont constitué eux-mêmes à base d’image segmentée à la main par un radiologiste afin d’entrainer et de valider leur modèle. Tout cela s’est fait sur des images pour lequel le foie était pré-segmenter. Inquiet du biais que cela pourrait introduire, j’ai donc posé la question au Dr Kadoury qui m’a assuré que ‘la segmentation du foie est assez précise. Elle va simplement fournir une région d’intérêt pour l’algorithme mais n’influencera pas l’apprentissage en amont’.
Le Dr kadoury et son équipe cherchait donc un moyen efficace de normaliser les images avant d’effectuer leur segmentation, ce qui semblait pouvoir permettre d’améliorer le processus. L’idée de normalisation d’un dataset n’est pas nouvelle mais elle se faisait (et se fait encore dans de nombreux cas) à l’aide de différentes méthodes de traitement d’image tel que l’application de filtre ou des égalisations d’histogrammes. Lors de l’interview il m’a précisé que ‘Souvent c’est un peu une magie noire pour identifier quels sont les filtres qu’on doit appliquer pour améliorer notre image et qui vont permettre une augmentation de performance à la fin’ ce qui touche principalement à la non-efficacité du pipeline traditionnel de traitement d’image. Ce dernier contient en effet cette ‘étape fastidieuse’ de normalisation du Dataset. Par ailleurs les filtres et autres transformations appliquées aux images pour un cas, pourrait ne pas s’appliquer dans un autre.

Le Dr kadoury et son équipe voulait donc à travers de multiples applications dans cette article montrer l’effet bénéfique de la normalisation et de l’utilisation du pipeline qu’ils avaient développé. En effet en utilisant un convolutionnal neural network (CNN) comme éléments de pre-processing cela leur permet de ‘laisser un modèle apprendre automatiquement’ la normalisation d’une image. Ce modèle pourra être entrainer pour chacune des applications afin d’effectuer la normalisation des images et d’améliorer les résultats de segmentation sans demander à l’équipe de créer un pipeline de pre-processing différents à chaque fois. Des méthodes quelque peu similaire, voir plus efficace ont vu le jour depuis, mais elles restent cantonné au images naturel. En effet on retrouve des modèle apprenant à supprimer differents type de bruit, ce qui serait plus efficace qu’une normalisation globale. Ce type d’application ne s’est pas encore transférer aux images médicale.
Bien que relativement efficace, ces découvertes et leurs applications en sont encore à l’étape de preuve de concept et ne se retrouveront pas de suite dans les hôpitaux. Il faudra en effet prouver son efficacité sur une large population afin de pouvoir passer à son déploiement. Ce n’est évidemment pas le seul projet visant à simplifier le travail des médecin et de leur prise de décisions. Ces logiciels pourraient un jour être déployés dans les hopitaux et aider les patients et les médecin en limitant l’erreur humaine également.
__________________________________________________________________________________________________
References :
[1] Drozdzal et al., “Learning Normalized Inputs for Iterative Estimation in Medical Image Segmentation.”
[2] Netgen, “Produits de contraste hépato-spécifiques en imagerie par résonance magnétique.”
[3] “Principe de l’IRM – Les agents de contraste.” Culture-chimie ENS.